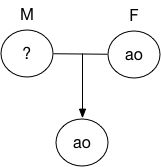
サンプリングによって先祖(親)の血液型を推定する
このページでは、家系図から親や先祖の血液型を推定する方法について考える。
母親の遺伝子型を推定する
親子の血液型の遺伝を考えてみよう。 遺伝子型を小文字のa,b,oで表すことにした場合、 父親の血液の遺伝子型がaoで、その子の遺伝子型もaoであったとする。一方、母親の遺伝子型は不明であったとしよう。 既知の情報から、母親の血液の遺伝子型を推定するには、どのように考えるのが良いだろうか。
ここで、遺伝子型を$x,y$として、父親の遺伝子型を$\textrm{F:}xy$、母親を$\textrm{M:}xy$、子供を$\textrm{C:}xy$のように表すとする。 ベイズの公式によれば、母親の遺伝子型を$xy$とすると、 $$ P( \textrm{M:}xy, \textrm{F:ao} | \textrm{C:ao} ) = \frac{P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:ao}) P(\textrm{M:}xy) P(\textrm{F:ao})} {\sum_{x,y,w,z} P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:}wz) P(\textrm{M:}xy) P(\textrm{F:}wz)} $$ となるが、いま、父親と子供の遺伝子型は完全に分かっているのだから、 $$ P(\textrm{F:ao}) = P(\textrm{C:ao}) = 1 $$ で、それ以外の遺伝子型についての確率は0であるから($P(\textrm{F:aa})=P(\textrm{F:ab})=\cdots=0$)、 $$ P( \textrm{M:}xy, \textrm{F:ao} | \textrm{C:ao} ) = \frac{P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:ao}) P(\textrm{M:}xy)} {\sum_{x,y} P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:ao}) P(\textrm{M:}xy)} $$ となる。 すると、子供がao型、父親もao型で、かつ、母親の遺伝子型が$xy$である確率は $$ \begin{eqnarray} P( \textrm{M:}xy, \textrm{F:ao}, \textrm{C:ao} ) = P( \textrm{M:}xy, \textrm{F:ao} | \textrm{C:ao} ) P(\textrm{C:ao}) \\ = \frac{P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:ao}) P(\textrm{M:}xy)} {\sum_{x,y} P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:ao}) P(\textrm{M:}xy)} \end{eqnarray} $$ である。
実際に値を求めるには、確率分布 $P(xy)$ を知らねばならないが、ここで、日本人の 遺伝子型の比率(a: 0.28, b:0.17, o:0.55)を想定し、 ハーディー・ワインベルクの法則を仮定すると、 以下のコードで母親の遺伝子型毎の確率が計算できる:
# coding: utf-8
ptab = {'a': 0.28, 'b':0.17, 'o':0.55}
gt_ALL = ['aa','ao','ab','bb','bo','oo']
def pt(gt):
x = gt[0]
y = gt[1]
if x==y:
return ptab[x]*ptab[y]
else:
return 2*ptab[x]*ptab[y]
# P(C|M,F)
def pc(gt_c, gt_m, gt_f):
gt=[ ]
for c1 in gt_m:
for c2 in gt_f:
gt.append(''.join(sorted(c1+c2)))
gt_c = ''.join(sorted(gt_c))
n_match=0
for c2 in gt:
if c2 == gt_c:
n_match += 1
return n_match/len(gt)
# メイン部
for t_m in gt_ALL:
psum=0
for x in gt_ALL:
psum += pc('ao',x,'ao')*pt(x)
p = pc('ao', t_m, 'ao')*pt(t_m) / psum
print(t_m,p)
血液型しかわからない場合
通常は、遺伝子型ではなくて血液型(A,B,AB,O)しかわからない。 そのような部分情報から、親(ここでは母)の血液型を推定する方法についても考えてみよう。
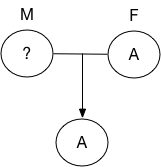
上図の例では、父親は aa, ao の可能性があり、他方、子供も aa, ao の可能性がある。 そうすると、母親は aa, ab, ao, bo, oo の可能性がある。
仮に、父親が aa で 母親が ao であれば、子供の遺伝子型が$xy$である確率は $$ P( \textrm{C:}xy | \textrm{M:ao}, \textrm{F:aa}) $$ 具体的には、子供が aa の確率は 1/2、aoである確率は 1/2である。
もし、父親が aa で子供が ao である場合は、母親が遺伝子型 $xy$ である確率は、前節のとおり $$ P( \textrm{M:}xy, \textrm{F:aa} | \textrm{C:ao} ) = \frac{P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:aa}) P(\textrm{M:}xy)} {\sum_{x,y} P( \textrm{C:ao} | \textrm{M:}xy, \textrm{F:aa}) P(\textrm{M:}xy)} $$ となる。 ここで、$P(\textrm{M:}xy)$ はハーディー・ワインベルクの法則によって与えるのが自然であろう。
今度は、もし母親が ao で、子供も ao であったとすると、父親の遺伝子型が$xy$である確率は $$ P( \textrm{M:ao}, \textrm{F:}xy | \textrm{C:ao} ) = \frac{P( \textrm{C:ao} | \textrm{M:ao}, \textrm{F:}xy) P(\textrm{F:}xy)} {\sum_{x,y} P( \textrm{C:ao} | \textrm{M:ao}, \textrm{F:}xy) P(\textrm{F:}xy)} $$ で与えられる。ここでも $P(\textrm{F:}xy)$はハーディー・ワインベルクの法則で決めることができるが、 実際に$xy$として許されるのは aa と ao のみである(父親の血液型がAであるから)。
このように、ある1人に着目すると、その「周り」の遺伝子型が既知であったとすれば、その人の遺伝子型の確率分布を推定するのは(式を計算する手間を除けば)容易である。
そこで、とりあえずの状態を設定しておいて、ある人に注目し、とりあえずの状態を元にして求めた確率に応じて、注目している人の状態を更新する作業を繰り返すようにする。 着目する人は順番に変えても、ランダムに決めてもよい(ただし、一様な重みでサンプルする必要がある)。すると、例えば、その状態は aa → aa → ao → aa → ... といった具合に目まぐるしく変化するはずである。
うまいことに、こうした更新作業を多数回繰り返すと、aaやaoの出現頻度は一定値に収束し、ちょうどそれぞれの遺伝子型の期待値(推定値)となることがわかっている。 このことは、父親と母親についても同様である。対象のうちのひとつ(ひとり)を選びながら、その状態を確率的に更新することで、知りたい量の期待値を得るこの方法は、 一般に、ギブスサンプリングと呼ばれている。
上記の手続きを100回繰り返すPythonコードの例を以下に示した:
右のコード中、関数 choose() で、NumPyの関数 np.random.choice() を使用している。
np.random.choice(選択肢のリスト, p=選択確率のリスト) とすると、リストで与えられた確率に応じて、選択肢のリストから要素が1つ返される。
# coding: utf-8
import random
import numpy as np
ptab = {'a': 0.28, 'b':0.17, 'o':0.55}
bt_A = ['aa','ao']
bt_AB = ['ab']
bt_B = ['bb','bo']
bt_O = ['oo']
gt_ALL = ['aa','ao','ab','bb','bo','oo']
def pt(gt):
x = gt[0]
y = gt[1]
if x==y:
return ptab[x]*ptab[y]
else:
return 2*ptab[x]*ptab[y]
# P(C|M,F)
def pc(gt_c, gt_m, gt_f):
gt=[ ]
for c1 in gt_m:
for c2 in gt_f:
gt.append(''.join(sorted(c1+c2)))
gt_c = ''.join(sorted(gt_c))
n_match=0
for c2 in gt:
if c2 == gt_c:
n_match += 1
return n_match/len(gt)
# P(M,F|C)
def pp(gt_m, gt_f, gt_c):
psum=0
for x in gt_ALL:
for w in gt_ALL:
psum += pc(gt_c,x,w)*pt(x)*pt(w)
return pc(gt_c, gt_m, gt_f)*pt(gt_m)*pt(gt_f) / psum
def choose(choices):
vtab=[]
ktab=[]
for k,v in choices.items():
vtab.append(v)
ktab.append(k)
plist = np.array(vtab)/sum(vtab)
return np.random.choice(ktab,p=plist)
# メイン部
state = {'M':'aa', 'F':'aa', 'C':'aa'}
for _ in range(100):
# 母親を更新
t_f = state['F']
t_c = state['C']
choices={ }
for t_m in gt_ALL:
choices[t_m] = pp(t_m, t_f, t_c)
state['M'] = choose(choices)
# 父親を更新
t_m = state['M']
t_c = state['C']
choices={ }
for t_f in bt_A:
choices[t_f] = pp(t_m, t_f, t_c)
state['F'] = choose(choices)
# 子供を更新
t_f = state['F']
t_m = state['M']
choices={ }
for t_c in bt_A:
choices[t_c] = pc(t_c, t_m, t_f)
state['C'] = choose(choices)
print(state)
 練習:ギブスサンプリングによる推定
練習:ギブスサンプリングによる推定
上記のコードの例を出発点に、母親の血液型を推定し、確率として表示するコードを書いてみなさい。
 ヒント
ヒント
確からしい値を得るには、サンプリングは十分な回数行う(長い系列を得る)必要がある。 また、サンプリングを始めてから暫くは状態が「安定」しないので、初期の状態は期待値の計算には用いないないようにすると良い。